高通量測序已經越來越多的用于生物學的研究。這些海量的數據中包含多種可用于進化和保護研究的分子標記。其中,微衛星分子標記由于其多態性較高,易于擴增等特點,廣泛的用于種群及保護遺傳研究中。
成都生物研究所曾曉茂研究員團組成員夏云博士,針對兩棲類微衛星跨物種擴增困難,而高通量測序相對便利的背景下,探討了不同來源,不同拼接方法,測序深度等對微衛星篩選結果的影響。對利用高通量數據篩選微衛星標記提供了綜合性的評估。研究發現,微衛星篩選的數量隨著數據量及讀長的增加而增加。數據拼接的策略會顯著影響微衛星的篩選結果,即K值越大,產生的微衛星數量少,但這些位點的重復次數多,暗示了更高的多態性。另外,轉錄組得到的微衛星位點相對于基因組位點顯示出較低的多態性但更易于與功能位點相關。
研究結果近期以題為“Microsatellite development from genome skimming and transcriptome sequencing: comparison of strategies and lessons from frog species”發表在國際基因組學期刊《BMC Genomics》上。
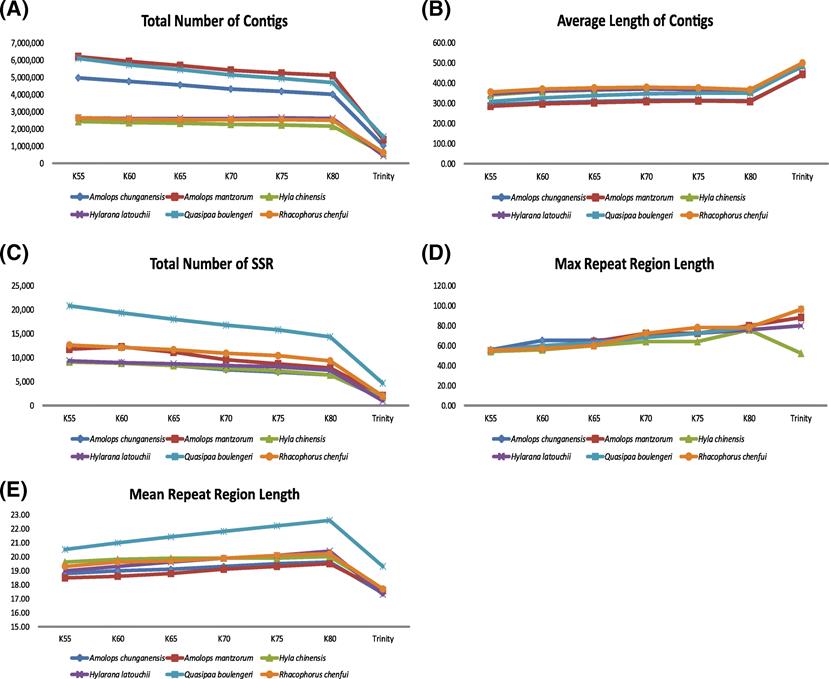
六種蛙類基因組不同拼接策略得到的微衛星位點差異
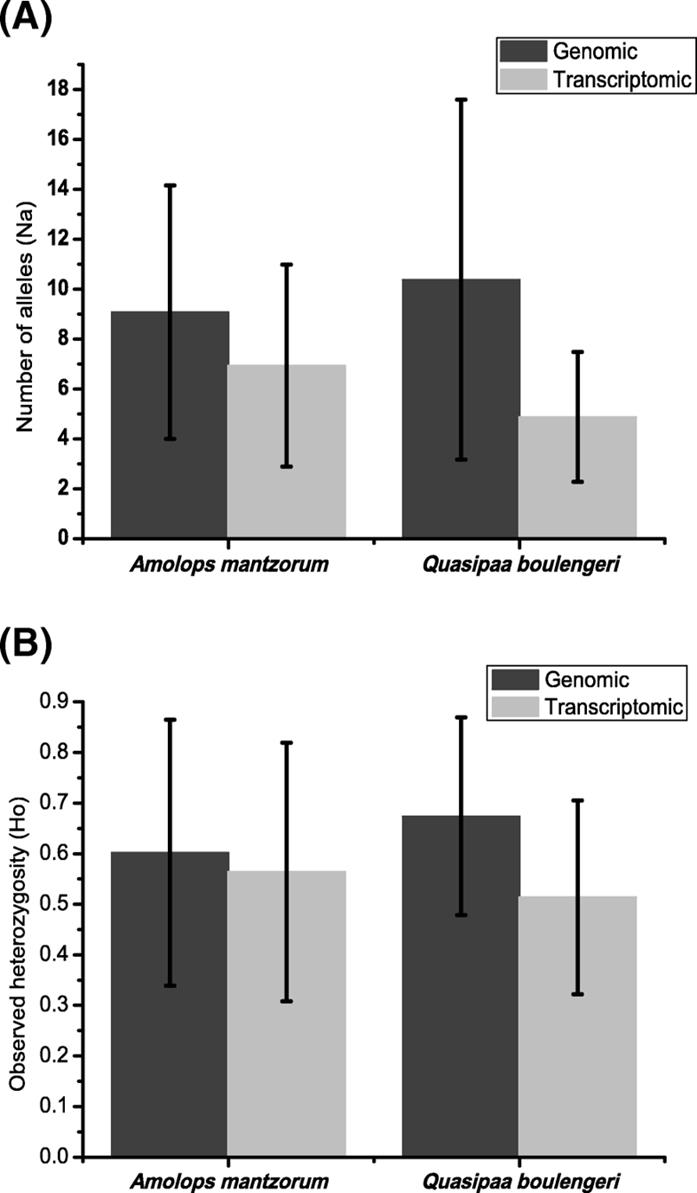
轉錄組和基因組篩選得到的微衛星多態性差異比較
